ТЕХНОЛОГИИ ТРЕХМЕРНОЙ НАУЧНОЙ ВИЗУАЛИЗАЦИИ И ГЕОМЕТРИЧЕСКОГО МОДЕЛИРОВАНИЯ В ЦИФРОВОЙ БИОМЕДИЦИНЕ
В.Е. Турлапов, Н.И. Гаврилов
Нижегородский государственный университет им. Н.И. Лобачевского, Россия
vadim.turlapov@cs.vmk.unn.ru, gavrilov86@gmail.com
Содержание
2. Обзор существующих разработок
2.1. Высокопроизводительная 3D визуализация и сегментация томограмм и микрофотографий
2.2. Проблема качества 3D визуализации
2.3. Параметризованное моделирование
2.4. Трехмерная сегментация, геометрическая реконструкция и анализ органов и человека в целом
3. Заключение: Актуальные проблемы и направления
Аннотация
Целью данной работы является обзор сегодняшнего уровня развития программного обеспечения трехмерной научной визуализации, сегментации и геометрического моделирования, обслуживающего задачи трехмерной цифровой биомедицины. Трехмерная цифровая биомедицина является интенсивно развивающейся отраслью, требующей вовлечения все большего числа специалистов. Актуальность темы обусловлена необходимостью формирования у научного сообщества научной визуализации целостного взгляда на эту область, видения актуальных задач и подходов к их решению.
Представлен арсенал программных комплексов, библиотек и систем, как ресурс медицинской 3D визуализации, сегментации, геометрической реконструкции и функционального моделирования. Представлены новые 3D технологии в медицине, созданные на базе этих библиотек. Кратко охарактеризованы возможности открытых библиотек, программных комплексов и систем для научной визуализации и сегментации, таких как VTK, ITK, CMake, ParaView, сопровождаемых Kitware Inc., VisIt (поддерживаемой Lawrence Livermore National Laboratory), ITK-SNAP, а также обеспечения геометрического моделирования – CGAL, Open CASCADE и SALOME.
Упомянуты известные открытые библиотеки для сегментации и реконструкции клеток по данным электронной микроскопии (ЭМ), такие как комплексы ILASTIK и CellBlender, обеспечение моделирования живой клетки - Monte Carlo Cell, а также специальный комплекс VMTK для моделирования динамики кровотока в сердечно-сосудистой системе.
Представлены возможности открытых и проприетарных, мировых и российских современных медицинских 3D визуализаторов Fovia HDVR®, Inobitec, Multivox3d, InVols (UNN). Обсуждается проблема качества 3D визуализации томограмм в условиях принципиального отсутствия эталона и ее решение на основе синтеза эталонного изображения путем усреднения серии изображений со случайным стартом луча. Утверждается принципиальная необходимость предыинтегрирования и исследования методов 3D визуализации в осях «производительность-качество».
Обсуждаются возможности, тенденции и глубина параметризации современных моделей анатомии человека от ведущих мировых фирм Nhumi Technologies, Visible Body, Plasticboy store, Zygote Inc. Существующие продукты и технологии 3D реконструкции тела пациента с камеры BodyKIT от Body Labs, Itseez3d от Itseez. Возможности и задачи медицинских технологий «виртуальный анатомический стол» и их современные реализации от Anatomage Inc., Sectra и аналогичные функции, но уже реализованные как облачные сервисы в InVols (ННГУ).
В заключении обсуждаются актуальные вызовы биомедицины, в том числе: сегментация и количественная оценка 3D аномалий (автоматизация диагностики); проблемы параметризации и персонализации модели анатомии человека, путем экстракции параметров из персональной томограммы. Обозначен эффективный подход к решению этих задач на основе объединения алгоритмов сегментации с геометрической реконструкцией в форме параметризованных моделей.
Новизна состоит в системности раскрытия темы, постановке актуальных задач и рекомендациях по их эффективному решению.
Ключевые слова: научная визуализация, геометрическое моделирование, 3D цифровая биомедицина, проблемы, решения, обзор
Биомедицина — раздел медицины, изучающий организм человека с теоретических позиций. Она включает сведения и исследования общие медицине и фундаментальным биологическим наукам, таким, как биология, в том числе нейробиология, гистология, биохимия, анатомия, патология, физиология, генетика, эмбриология, биомедицинский инжиниринг, зоология, ботаника и микробиология. Биомедицина создает фундамент для всех медицинских приложений, диагностики и лечения, делает возможным внутриклеточное и молекулярное понимание механизмов болезни, появление новых лекарственных средств, стволовых клеток для клеточной терапии. На переживаемом нами уровне развития науки и технологий вектор развития цифровой биомедицины отчетливо направлен в сторону трехмерной биомедицины, как самой высокотехнологичной и инновационной. Обзор посвящен достижениям научной визуализации и геометрического моделирования, которые обеспечили современное развитие 3D биомедицины, ряду интересных результатов этого развития и проблемам, которые находятся в процессе решения и ожидают усилий со стороны сообщества компьютерной графики.
Вызовом для развития трехмерной цифровой биомедицины послужило взрывное развитие томографии и трехмерной электронной микроскопии, создающих последовательности двумерных слоев высокого разрешения. Быстро обнаружилось, что трехмерные данные способны качественно улучшить результаты диагностики и исследований, если будут созданы методы и программное обеспечение (ПО) 3D обработки и визуализации этих данных, и, на их основе, новые трехмерные технологии в медицине и биологии. В связи с этим перед компьютерной графикой и геометрическим моделированием возникли следующие задачи: 1) высокопроизводительная и, одновременно, качественная 3D визуализация и сегментация томограмм, хранимых в специальном медицинском формате DICOM; 2) параметризация геометрических моделей человека, его органов и систем, ориентированная на решение задачи персонализации модели; 3) трехмерная сегментация и геометрическая реконструкция органов и человека в целом по данным томографии и другим источникам; 4) детектирование аномалий и их количественная 2D-3D оценка (автоматизация диагностики). Разумеется геометрия и графика в биомедицине должны поддерживать и функциональное моделирование человека, его органов, систем и манипулирование ими так же, как это делается на этапах пред- и постпроцессинга в инженерных системах. Выходом такого функционального моделирования должны быть исследовательские и обучающие биомедицинские системы виртуальной реальности.
Пожалуй наиболее существенный вклад в создание программного обеспечения современной биомедицины внесла компания Kitware, Inc. (www.kitware.com, USA-1998). Она организовала создание целой серии Open Source программных продуктов, жизненно необходимых трехмерной цифровой биомедицине. Прежде всего это: 1) Visualization Toolkit (VTK, www.vtk.org) – программный комплекс для обработки изображений, полигональной 3D графики и научной визуализации; 2) Insight Segmentation and Registration Toolkit (ITK, www.itk.org) – программный комплекс для сегментации трехмерных медицинских данных, получивший в США статус национальной библиотеки программ для создания медицинских систем; 3) CMake – обеспечение сборки сложных систем, которое первоначально разрабатывалось для сборки ITK и ITK-приложений. CMake управляет процессом сборки используя простые платформо- и компиляторо-независимые конфигурационные файлы; 4) ParaView (www.paraview.org) - открытая система высокопроизводительной научно-технической визуализации, реализующая практически все существующие на сегодняшний день приемы научной визуализации. Имеет она и возможности медицинской 3D визуализации. Визуализация ParaView построена на библиотеках VTK и VTK-m – версии, ориентированной на использование графических процессоров. Сегодня уже существует web-вариант системы доступный на сайте www.paraview.org/web. Сегодня Kitware не только продолжает работать в Medical Computing, в HPC & Visualization и Software Process, но и расширила свои интересы на Computer Vision и Data & Analytics. Активными партнерами Kitware являются более 80 университетов и научных центров США.
Интересен и аналог ParaView открытая система научной визуализации VisIt (http://visit.llnl.gov/), созданная также в США в Lawrence Livermore National Laboratory. Эта система также базирована на VTK и VTK-m, также мультиплатформенная и интерактивная. Возможно в силу специфики создания она несколько более интересна физикам.
Десятилетний совместный проект лаборатории Penn Image Computing and Science Laboratory (PICSL, университета штата Пенсильвания) и института Scientific Computing and Imaging (SCI, университет штата Юта), поддержанный грантом National Institute of Biomedical Imaging and Bioengineering (NIBIB), закончился созданием компактного и сравнительно простого в использовании комплекса ITK-SNAP (www.itksnap.org). Комплекс ориентирован на интерактивную сегментацию с одновременной работой оператора в трех ортогональных проекциях и на использование в каждой из проекций возможностей метода активного контура (snake) развитых авторами. К настоящему времени пользователями ITK-SNAP в мире сделаны уже многие сотни публикаций.
В сфере 3D сегментации и визуальной реконструкции микрофотографий с электронных микроскопов (ЭМ), которая пока требует существенно большей доли ручной работы, создан открытый программный комплекс ILASTIK (http://ilastik.org), ориентированный на интерактивную классификацию, сегментацию и последующий анализ. Модульное ПО обеспечивает автоматизированную пиксельную и объектную классификацию, полуавтоматический трекинг объектов, полуавтоматическую сегментацию и подсчёт объектов без их детектирования.
ILASTIK разработан специальной командой университета Гейдельберга (Heidelberg Uni., Germany) при финансовой поддержке ряда сообществ и организаций: Heidelberg Collaboratory for Image Processing (HCI); HHMI (Howard Hughes Medical Institute) Janelia Farm Research Campus; CellNetworks Excellence Cluster (www.cellnetworks.uni-hd.de).
Интересны сайт и ресурс MMBioS (http://mmbios.org/) международного сообщества биологов, образовавшегося вокруг The National Center for Multiscale Modeling of Biological Systems (USA). Здесь в разделе Software предлагается к использованию несколько крупных программных комплексов, созданных международными усилиями. Наиболее известными из них являются CellBlender (MMBioS downloads page) и Monte Carlo Cell (http://mcell.org/).
В медицинской 3D визуализации результатов томографии есть и коммерческие программные комплексы. Самым известным из них остается продукт Fovia HDVR® (High Definition Volume Rendering) фирмы Fovia Inc. (California, USA, www.fovia.com), встроенный в ПО большей части зарубежных томографов (Рис.1). Существенной чертой этого продукта является отказ от использования графических процессоров.

Рис. 1. Сайт компании Fovia Inc., представляющий HDVR®
На российском рынке есть продукт фирмы Inobitec (www.inobitec.ru, Москва), позиционируемый авторами как отечественный аналог HDVR®. Этот продукт практически уступает HDVR® только производительностью.
Интересен также российский продукт известный сегодня как Автоматизированное Рабочее Место (АРМ) “Гамма Мультивокс Д2” (www.multivox.ru/multivox3d.shtml), предназначенное для работы с 2D/3D медицинскими изображениями и сериями изображений. Этот продукт зародился в МГУ им. М.В. Ломоносова и с самого начала был и остается ориентированным не столько на 3D визуализацию, сколько на 3D сегментацию и количественные оценки остро необходимые диагносту. Его выгодно выделяют такие функции как: полуавтоматическая 3D сегментация, геометрическая реконструкция поверхности, редактирование, измерение объемов и визуализация сегментированных объектов псевдоцветами с серой текстурой.
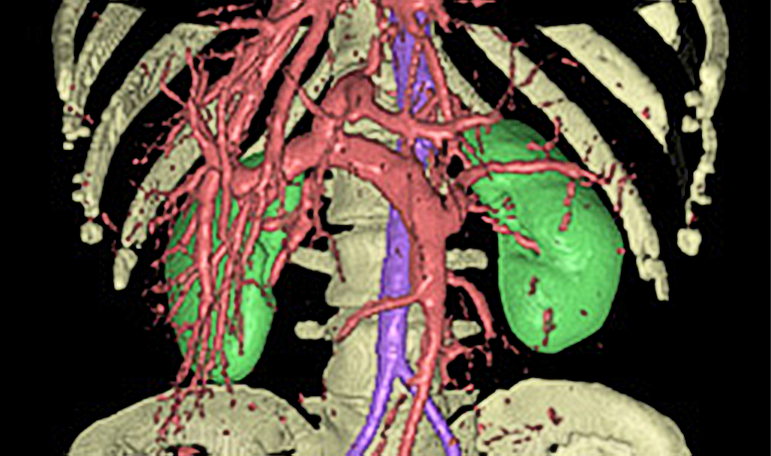
Рис. 2. Визуализация сегментированных и геометрически реконструированных сосудов печени в АРМ «Мультивокс»
Возможно рекордную на сегодняшний день производительность имеет открытый DICOM визуализатор InVols, разработанный в ННГУ им. Н.И. Лобачевского. Invols использует лучшие современные алгоритмы DVR, включая предварительное интегрирование и блочную декомпозицию, и оптимизирован под архитектуру GPU. Еще в апреле 2012 он был способен визуализировать в реальном времени томограмму из 5168 слоев: «GPU RayCasting for a huge dataset» (https://www.youtube.com/user/ngavrilov86). Производительность InVols продолжает с каждым годом расти вместе с производительностью GPU.
Несомненно, что на качество 3D визуализации влияют 3 главных фактора: 1) шум в исходных данных; 2) способ интерполяции при выборке значений на регулярной сетке данных при интегрировании вдоль луча; 3) постклассификация (классификация после интерполяции) выбранного значения посредством Transfer Function.
В последнее время много сделано для аппаратно-программной минимизации шума в каждом из 2D слоев томограммы. Сделан и серьезный шаг в удалении шума за счет использования когерентности соседних слоев томограммы [12]. Поэтому будем рассматривать оставшиеся 2 фактора.
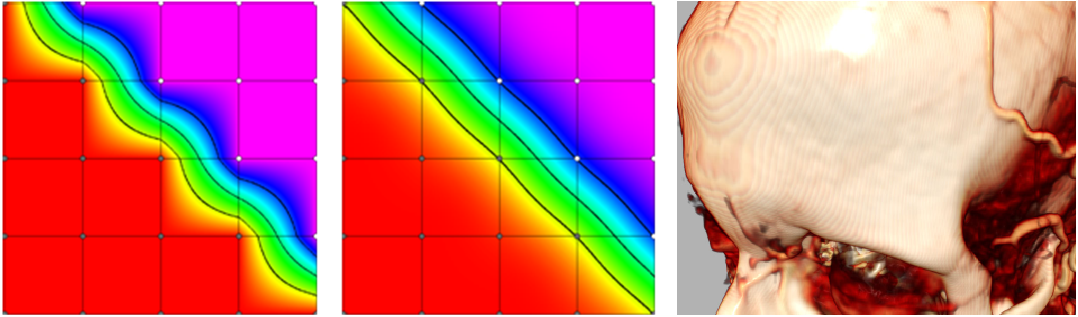
Рис. 3. Сравнение искажений, вносимых на модельной сетке данных билинейной интерполяцией (слева) и бикубическим сглаживанием (справа) и реальные артефакты трилинейной интерполяции в виде древесных колец
На рис.3 показано влияние способа интерполяции данных на качество изображения. Визуально так же проявляются и артефакты постклассификации. Понятно, что с дефектами интерполяции можно бороться либо путем увеличением разрешения томограммы, либо, например, применением трикубической интерполяции или даже фильтра Ланцоша. Блестящее решение в отношении артефактов постклассификации предложено авторами метода Pre-integrated Direct Volume Rendering (PDVR) [3]. Этот метод не только качественно улучшил синтезируемое изображение, но и сделал процесс интегририрования вдоль луча математически корректным. Более поздние методы адаптивного выбора шага peak finding and differential sampling (ASM) [4] и виртуальных выборок (VS) [5] пытались отказаться от PDVR по мотивам их более высокой производительности для некоторых данных. Однако, используя критерий производительности, они не контролировали качество изображений. Объективной причиной для этого является отсутствие эталона.
Проблема измерения и обеспечения заданного качества 3D визуализации томограммы решена приемлемым образом в работе [2]. Предложен метод измерения (количественной оценки) артефактов и качества 3D визуализации, не требующий эталонного изображения, и, следовательно, являющийся универсальным подходом к измерению качества реконструкции любым методом 3D визуализации. Метод основан на генерации эталонного изображения, как математического ожидания пикселей серии изображений, полученных путем зашумления старта луча в пределах одного шага вдоль луча. Для оценки отклонения от матожидания выбрана традиционная для сигналов и изображений величина PSNR в dB. Обсуждены условия корректности применения этой оценки. Предложен также метод Pre-integrated virtual sampling (PLVS, PCVS, см. рис.4-5), объединяющий достоинства виртуальных выборок с предынтегрированием.
Предложен новый подход к разработке алгоритмов 3D визуализации, основанный на исследовании метода в осях производительность-качество. Выполнено сравнительное исследование нескольких современных методов в осях производительность-качество (рис.4,5).
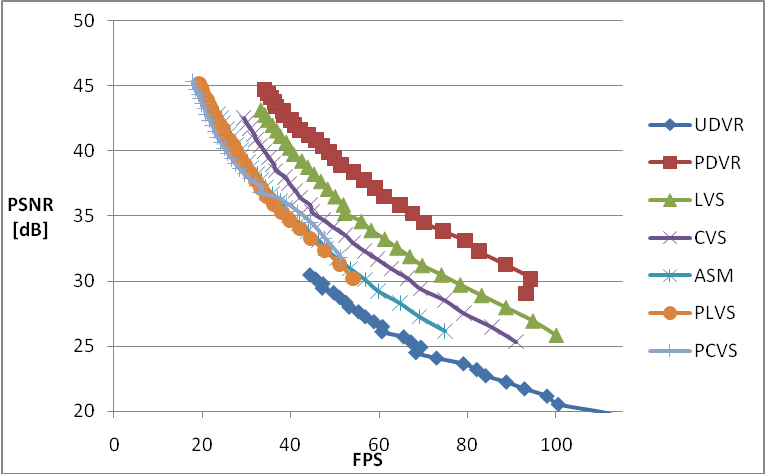
Рис. 4. Качество (PSNR, dB) / производительность (кадров/сек, FPS) для случая трилинейной фильтрации данных без освещения
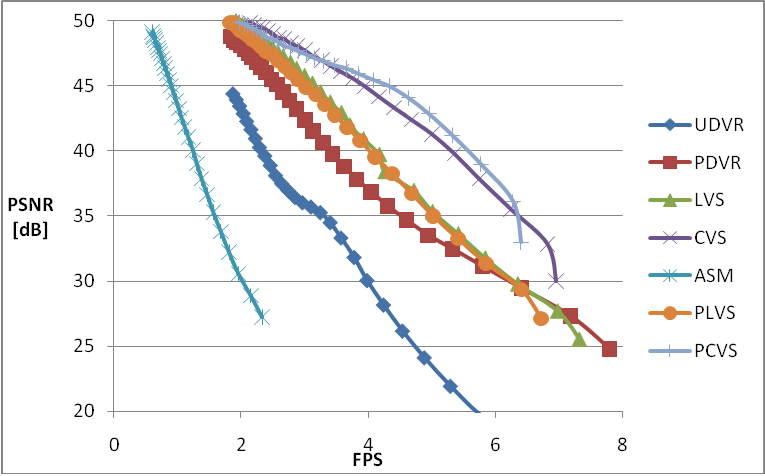
Рис. 5. Качество (PSNR, dB) / производительность (кадров/сек, FPS) для случая трикубической фильтрации данных с освещением.
Исследование проведено для методов в реализации авторов публикации [2]. Обозначения: UDVR – обычный Direct Volume Rendering; PDVR - Pre-integrated Direct Volume Rendering [3]; ASM - peak finding and differential sampling [4]; LVS, CVS – Virtual Sampling с линейной и кубической интерполяцией виртуальных выборок вдоль луча; PLVS, PCVS – Pre-integrated virtual sampling с линейной и кубической интерполяцией реальных выборок [2]. Маркеры вдоль кривых соответствуют линейному изменению частоты выборки значений вдоль луча от 1 до 8 выборок на воксел.
Хочется надеяться, что метод количественной оценки качества визуализации окажется полезным не только в биомедицинской визуализации, но и в вычислительной механике жидкости и газа, где актуально детектирование и качественная визуализация вихрей [10].
Линия трехмерного моделирования человека, его органов и систем, представлена достаточно большим числом фирм и комплексов моделей. Исторически абсолютное большинство 3D моделей были созданы для целей наглядного изучения анатомии и не являются параметризованными. Авторы наиболее продвинутых продуктов уже начали движение в сторону создания параметризованных моделей и полноты параметризации.
Лидер рынка США по цифровым моделям для обучения фирма Visible Body (1996, http://www.visiblebody.com/), ежегодно получающая премии за лучший продукт для обучения в области биомедицины, начала производить модели реализующие локальное движение.
Более молодая английская фирма Plasticboy (2005-2009) предлагает сегодня на своем сайте новую версию анатомических моделей Male-Female серии Anatomy Models Store, параметризованную для движения (rigged model) в среде Autodesk Maya: V07 Anatomy 3D models (Motionbuilder Compatible). Остальные форматы моделей (для других 12 систем 3D моделирования) поставляются стандартными (т.е. без движения). Примерная цена – около $2000 на 1 рабочее место. Пример на сайте компании демонстрирует работу при движении всего костно-мышечного каркаса близкую к естественной (http://www.plasticboy.co.uk/store и рис.6).

Рис. 6. Пример параметризации движения в 3D модели нормальной анатомии фирмы Plasticboy.
Признанным лидером в области 3D моделирования в биомедицине считается фирма Zygote Inc. (http://www.zygote.com/). С 1994 года она оказывает наукоемкие услуги по 3D моделированию в биомедицине, индустрии развлечений и на профессиональном рынке. Сегодня наряду с поверхностными полигональными текстурированными моделями в форматах систем 3d-моделирования: 3D Studio Max; Blender (OBJ); Cinema 4D; Generic OBJ; Maya (http://www.zygote.com/poly-models), предлагаются также твердотельные CAD-модели для использования в биоинженерных нуждах внутри CAD систем: ProE/Creo; IGES; ParaSolid; SolidWorks; Step. Рыночная цена пары поверхностных моделей Male-Female – около $25000. Чем выше степень параметризации модели, тем технологичнее ее применение на указанных рынках.
Именно на основе таких моделей в 2014 возникла фирма EZ (Germany). Сегодня она предлагает анатомические 3D препараты, отпечатанные на 3D принтере (Printed Anatomy Series, www.3danatomyseries.com) и, возможно, будет затем осваивать и область протезирования.
Все эти модели созданы как благодаря развитым коммерческим системам 3D моделирования, так и современным открытым библиотекам и средам геометрического моделирования. К числу таких открытых библиотек следует отнести прежде всего CGAL - The Computational Geometry Algorithms Library (www.cgal.org) [7]. Проект по созданию этой библиотеки стартовал в 1996 году как международный европейский проект первоначально 7 сторон: Utrecht University (The Netherlands, prime contractor), ETH Zürich (Switzerland), Free University of Berlin (Germany), INRIA (France), Max-Planck-Institute Saarbrucken (Germany), RISC Linz (Austria), Tel-Aviv University (Israel), и финансировался несколькими крупными европейскими научными программами (GALIA, ECG, ACS, Aim@Shape). С 2003 года стали доступны коммерческие лицензии CGAL. CGAL C++ library, заканчиваясь построением сеток, обладает функциональной полнотой и широко используется для создания прикладных систем с геометрическим базисом в следующих областях: GIS, CAD, молекулярная биология, медицина, компьютерная графика, робототехника и т.д.
Отечественным аналогом CGAL является геометрическое ядро C3D (http://c3dlabs.com), разрабатывавшееся с 1995 в интересах отечественной CAD КОМПАС-3D компании АСКОН. В ноябре 2012 C3D Labs стала самостоятельной компанией, работающей на интернациональный рынок.
Близким по функциональности к CGAL проектом, но с чисто французским началом, является открытая система и технология для геометрического проектирования и инженерного анализа (Open CASCADE Technology, http://www.opencascade.org/). CAS.CADE является аббревиатурой от названия программного комплекса Computer Aided Software for Computer Aided Design and Engineering, который был разработан в начале 1990-х французской компанией Matra Datavision, разработавшей в1980 CAD Euclid. CAS.CADE стал затем основой для такой известной системы как CATIA. В 1999 Matra Datavision сделала CAS.CADE открытой платформой.
На основе данной библиотеки построена открытая система трехмерного моделирования, сеточной декомпозиции и инженерного анализа SALOME (www.salome-platform.org), способная обеспечить платформу пред- и пост-обработки для численного моделирования, как в инженерных приложениях, так и в медицине.
Параллельно созданы и более скромные открытые библиотеки, и подобные коммерческие, как например, The Geometric Modelling Library (GML), созданная международной компанией Renishaw (http://www.renishaw.com/geometricmodelling/en/the-gml--14749).
На такой мощной компьютерно-геометрической поддержке появились, как отдельные прикладные модели, так и целые программные комплексы (libraries and tools), обеспечивающие функциональное моделирование отдельных органов и систем организма человека. Одним из известных комплексов является VMTK (www.vmtk.org) - the Vascular Modelling Toolkit. VMTK решает задачи 3D реконструкции, геометрического анализа, генерации конечно-элементных сеток, анализа поверхностей и моделирования кровотока в сердечно-сосудистой системе человека.
Развитие систем 3D моделирования сделало возможным использование 3D модели пациента, как основы для интегрирования и совместной трактовки результатов его исследования современными методами медицинской диагностики. Исследования и создание систем в этом направлении активно поддерживается компанией IBM, мировым лидером производства суперЭВМ, увидевшей здесь перспективы для массового применения суперЭВМ в реализации технологий на основе больших данных. Результатом завершения проекта IBM 3D Avatar стали три новых технологии, поддерживаемых новой фирмой Nhumi Technologies (2009, www.nhumi.com, читается как New Me). По смыслу – это новый трехмерный Я, построенный от начала до конца на трехмерном образе пациента (3D-аватаре конкретного человека), его медицинских исследований, 3D планировании и наблюдении процесса лечения. Первая технология Study Insight – сервис интеграции данных различных исследований пациента с возможностью коллективного Интернет-доступа к ним врачей и проведения телеконсилиумов по пациенту. Технология Visual Health – сервис навигации по 3D модели анатомии человека (около 3000 объектов) для врачей с любого стандартного web-клиента с привязкой клинических данных к органам, образно говоря, - трехмерная медицинская карта пациента (Рис.7).
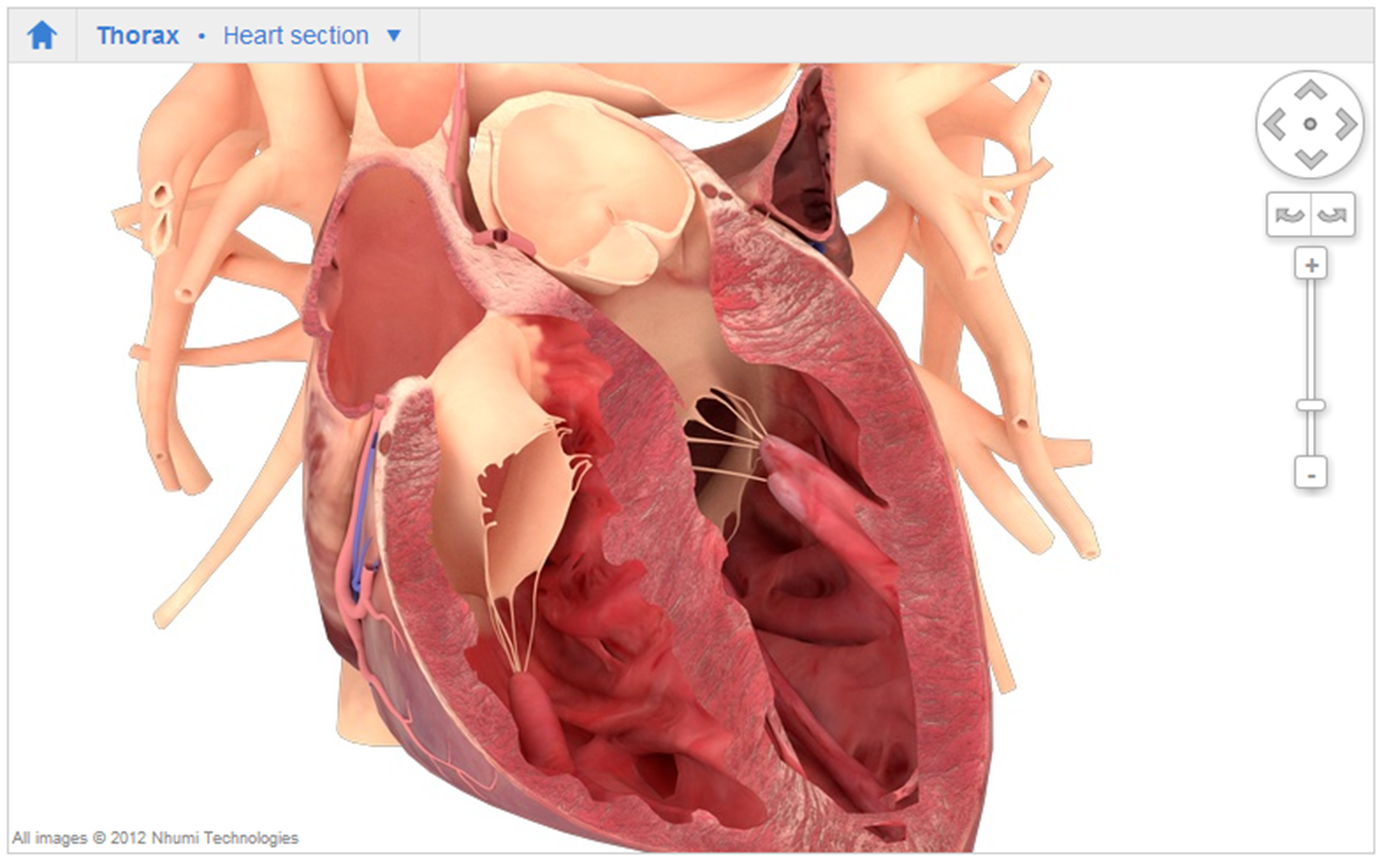
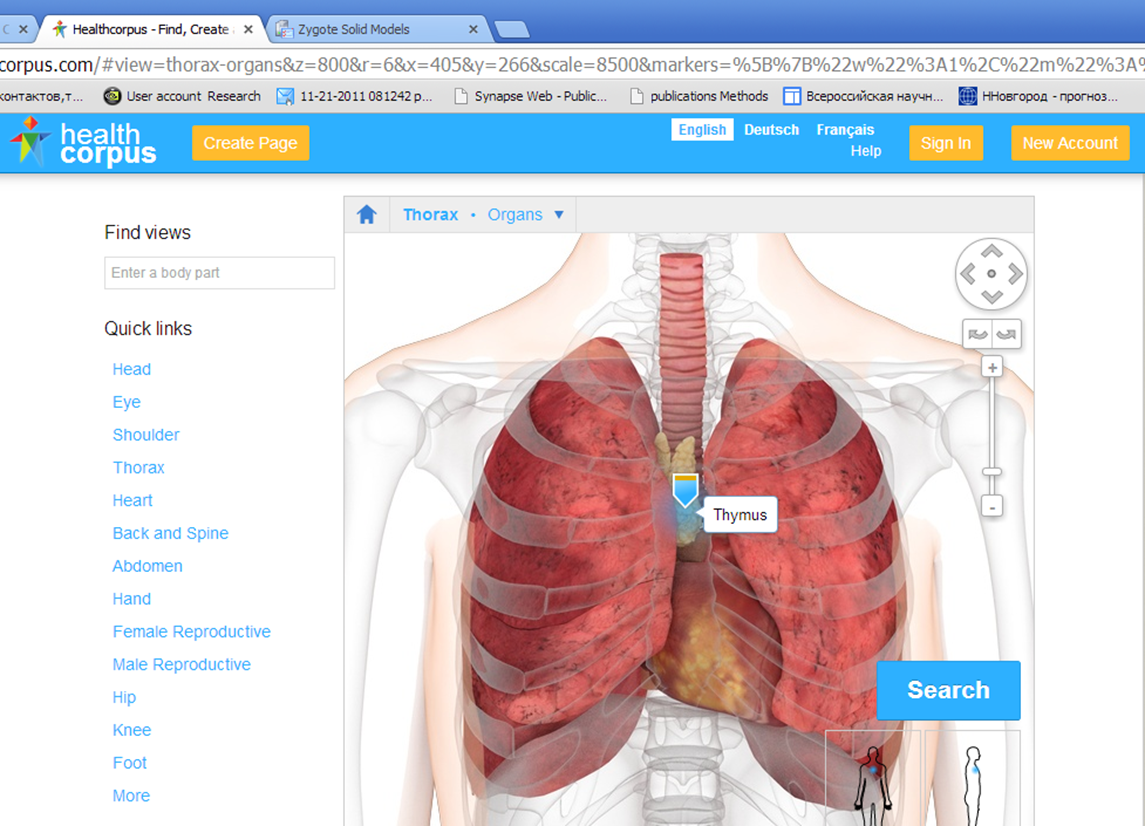
Рис. 7. Примеры моделей, используемых в системах Visual Health и Healthcorpus
Как видим, даже общая трехмерная модель анатомии начинает играть существенную роль в медицинских системах. Тем более - персональная модель анатомии конкретного пациента. С ее построением с еще большим основанием можно говорить о персональной медицине. Имеет смысл начинать прием врача с 3D реконструкции тела пациента. Наиболее дешевый способ для этого – использование изображений с видеокамер. Можно говорить, что эта задача уже подхвачена специалистами и нашла неплохие решения. Одно из решений можно увидеть на сайте фирмы Body Labs (www.bodylabs.com) – подготовлена бета-версия продукта BodyKIT для высокоточной реконструкции 3D модели тела и захвата его движения с MS Kinect. В проекте также декларированы возможности провести сравнение, морфинг, анимацию, и усреднение тел. Другое решение – на сайте компании Itseez, поддерживающей и развивающей всемирно известную библиотеку компьютерного зрения OpenCV, (http://itseez.com/products/itseez3d/) – уже работающее ПО 3D реконструкции тела человека и других объектов (Рис.8).

Рис. 8. Пример 3D реконструкции на сайте Itseez
Для медицинских технологий, использующих задачи 3 и 4, найдено интересное прикладное решение, получившее название Виртуальный анатомический стол (Dissection Table). Впервые такой стол был передан в Стэндфордский университет (Stanford Uni.) фирмой Anatomage Inc. (www.anatomage.com) в 2011 для экспериментального использования и развития. Главной идеей стола было соединение 3D визуализации индивидуальных данных томографии с параметризованной моделью анатомии (Рис.9).

Рис. 9. Пример, представленный на сайте http://medical.anatomage.com, объединения полигональной 3D модели (внутренних органов и кровеносной системы) с воксельной реконструкцией скелета и контура тела, извлеченных из реальной томограммы
Это предлагалось использовать прежде всего для целей обучения студентов. К объектам модели можно легко привязать любую обучающую информацию, а конкретная томограмма должна давать информацию о реальном состоянии и патологиях органов. Однако универсальная реализация данной концепции за пределами частного случая, представленного на сайте оказалась слишком трудоемкой и не завершена до настоящего времени. Развитие системы в прошедшие 4 года пошло по линии накопления библиотеки распространенных патологий органов (к настоящему времени в системе Anatomage Table собрано около 120 патологий), и накопления инструментария сегментации, геометрической реконструкции и методов анализа отдельных частей тела. Этот программный инструментарий получил название Invivo (http://medical.anatomage.com/medical-products/invivo5). На сегодняшний день он содержит 8 функций и сертифицирован для применения в медицине. Каждый экземпляр Anatomage Table поставляется с пакетом Invivo.
Более простой и прагматичный продукт Sectra Visualization Table предложен в 2012 году шведской фирмой Sectra (http://www.sectra.com/medical/sectra_table/), которая уже более 20 лет работает на рынке медицинской визуализации. Это стол для 3D визуализации томограмм в реальном времени, который позиционируется как полезный для предоперационной подготовки хирурга, коллективных обсуждений результатов томографии и изучения патологий студентами на реальном материале томограмм. Программное обеспечение 3D визуализации поставляется вместе со столом. В 2015 году для владельцев Sectra Table появился облачный вариант визуализации на планшете.
В России также в настоящее время завершается разработка виртуального стола более подобного Anatomage Table. Она выполняется силами малого предприятия при Саратовском медицинском университете на основе разработанного там атласа нормальной и патологической анатомии человеческого тела InBody Anatomy (http://inbody.pro, http://www.npo-leader.com/) с более чем 100 патологиями.
В нижегородском университете в 2013 году реализован облачный сервис высокопроизводительной 2D-3D визуализации томограмм для организации на его основе персональных кабинетов врача и пациента. Экспериментальный вариант сервиса (http://85.143.3.10:4526) способен работать также в режиме виртуального стола типа Sectra Table (Рис.10).
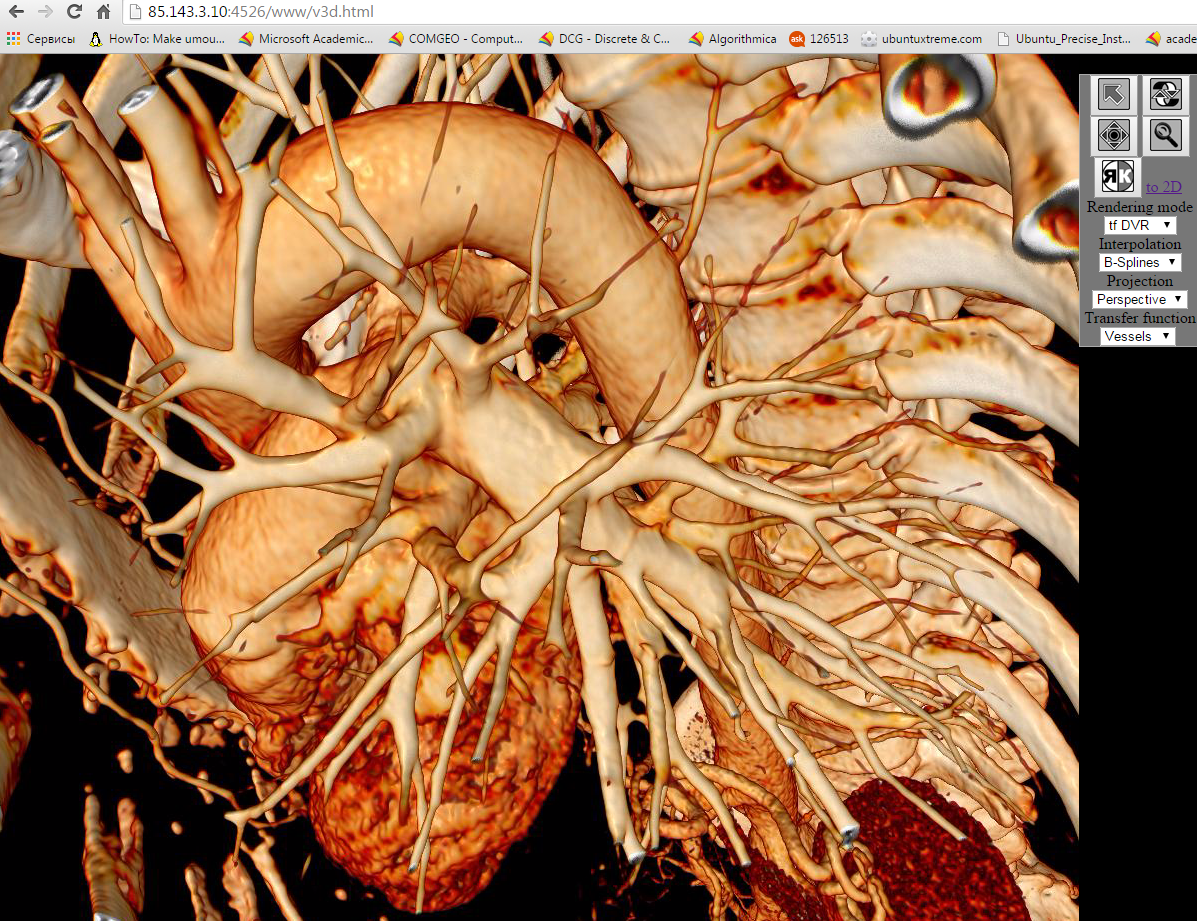
Рис. 10. Пример 3D визуализации средствами облачного сервиса ННГУ
Ближайшие задачи. Отражение части решаемых в настоящее время проблем можно найти среди новых функций Invivo5 на (http://medical.anatomage.com/medical-products/invivo5). Это наполнение и расширение задач: Compare Cases - попарное сравнение случаев (количественное детектирование динамики), причем не только в 2D, но и в 3D (например, сравнение объема легких на вдохе и выдохе). Это также Isolate Anatomy and Capture Videos – пользователь может реконструировать отдельную деталь скелета и выполнить с ней различные манипуляции: масштабировать, повернуть, параллельно перенести. Еще шире возможности опционального компонента Medical Design (MD), который может как геометрически реконструировать фрагменты томограммы (Create Digital Models from Patient Scans), так и импортировать в созданные модели внешние объекты, например, искусственный клапан - в сердце.
Направление развития алгоритмов сегментации и реконструкции. Очевидно, реконструкция выполняется на основе сегментации, а ее возможности сегодня крайне ограничены и нуждаются в дальнейшем развитии и регуляризации. Особенно сложна сегментация и диагностика мягких тканей. Полноценное детектирование аномалий органа должно начинаться с точного детектирования его границ. Что чаще всего крайне сложно сделать по данным томограммы из-за того, что томограф имеет ограниченное разрешение, и при шаге срезов в 0.5 мм не может достоверно детектировать объекты величиной или толщиной (для тонких границ) меньше 1 мм. Эта проблема вплотную соприкасается с проблемой слияния 3D модели нормальной анатомии с томограммой тела пациента или фрагмента тела и экстракции параметров модели из персональной томограммы. Однако, здесь возможно движение навстречу друг-другу со стороны углубления и специализации параметризации 3D модели и со стороны методов сегментации. Серьезным шагом в этом направлении является работа Computational Bodybuilding [6], представленная на SIGGRAPH’2015. Есть надежда на регуляризацию алгоритмов сегментации за счет применения таких опорных моделей. Этот же путь представляется эффективным и для создания высокопроизводительных алгоритмов сегментации и реконструкции.
Создание функциональных (динамических) персональных моделей. Есть также важнейшая задача полноценного вовлечения ультразвуковой диагностики в 3D реконструкцию и задача реконструкции на этой основе не статических, а динамических (особенно допплеровских) объемных моделей: во многих случаях исследования динамики органа/системы ультразвуковое исследование остается золотым стандартом. Примером реконструкции кровотока в сердце по данным ультразвукового исследования является работа [8]. Также актуальна обработка томограмм снятых в динамике, адекватная по производительности так называемым «быстрым» МРТ томографам (FLASH MRI), появляющимся на рынке.
Довольно широким фронтом ведутся работы по функциональному моделированию отдельных органов и систем тела человека с перспективой создания «виртуального клона человека», по журналистской терминологии. Примерами таких работ являются недавние работы по реконструкция кровотока: в сосудах ног [1]; прогноза хирургического лечения аневризмы артерии мозга [9] и реконструкция мелких сосудов глазного дна [13]. Ведутся работы по электро-механическому моделированию сердца [11]. Во всех этих случаях за компьютерной графикой остается геометрографическое сопровождение функционального моделирования персонального виртуального организма. Свежим примером такой функции является визуализация динамики сокращающегося киберсердца пациента (https://youtu.be/2LPboySOSvo), построенной в RIKEN на японском суперкомпьютере К и представленной впервые 04 июня 2015 на конференции SIGGRAPH 2015 (рис. 11).
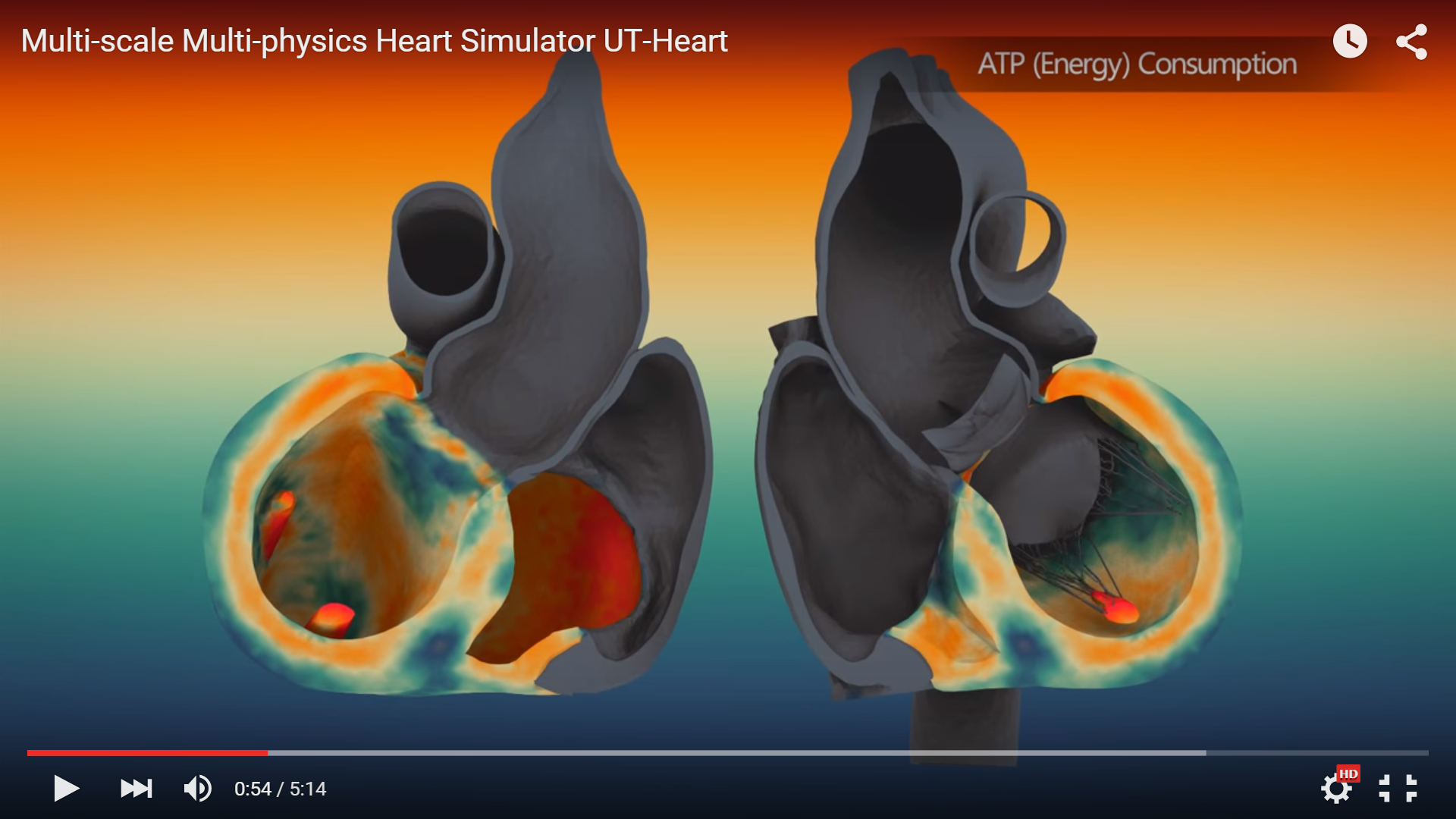
Рис. 11. Визуализация динамики сокращающегося киберсердца пациента (https://youtu.be/2LPboySOSvo)
Работа поддержана грантом (соглашение от 27 августа 2013 г № 02.В.49.21.0003 между МО РФ и ННГУ)
1. Gamilov T., Ivanov Yu., Kopylov P., Simakov S., Vassilevski Yu. Patient specific hemodynamic modeling after occlusion treatment in leg. Mathematical Modelling of Natural Phenomena, 2014, vol. 9, no. 6, pp. 85-97
2. Gavrilov, N. I.; Turlapov, V. E. Novel approach to development of direct volume rendering algorithms based on visualization quality assessment. Programming and Computer Software, 2014, vol. 40, no. 4, pp. 174-184
3. Engel K., Kraus M., Ertl T. High-quality pre-integrated volume rendering using hard-ware-accelerated pixel shading. Proc. of Graphics Hardware 2001, pp. 9-16
4. Knoll A., Hijazi Y., Westerteiger R., Schott M., Hansen C., Hagen H. Volume ray casting with peak finding and differential sampling. IEEE Transactions on Visualization and Computer Graphics, 2009, vol. 15, no. 6, pp. 1571–1578.
5. Lee, B., Yun, J., Seo, J., Shim, B., Shin, Y. G., & Kim, B. Fast high-quality volume ray casting with virtual samplings. IEEE Transactions on Visualization and Computer Graphics, 2010, vol. 16, no. 6, pp. 1525-1532.
6. Saito Sh., Zhou Zi-Ye, Kavan L. Computational Bodybuilding: Anatomically-based Modeling of Human Body. ACM Transactions on Graphics, 2015, vol. 34, no. 4, Article 41
7. The CGAL Project. CGAL User and Reference Manual. CGAL Editorial Board, 4.6.2 edition, 2015.
8. Yatchenko A., Krylov A. Cross-Frame Ultrasonic Color Doppler Flow Heart Image Unwrapping. Lecture Notes in Computer Science, 2015, vol. 9126, pp. 265−272
9. Yatchenko A.M., Gavrilov A.V., Boldyreva E.V., Arkhipov I.V., Grigorieva E.V., Gudkov I.M., Krylov V.V. Blood Flow Prediction and Visualization within the Aneurysm of the Middle Cerebral Artery after Surgical Treatment. Proceedings of IMTA-5, 2015, Portugal, pp. 108-113
10. Бондарев А., Галактионов В. Современные направления развития визуализации данных в вычислительной механике жидкости и газа. Научная визуализация, 2013, т. 5, № 4, с. 18 - 30
11. Костин В.А., Осипов Г.В. Самосогласованная электромеханическая активность в упруго закрепленном волокне сердечной мышцы. Проблемы прочности и пластичности, 2014, т. 76, № 4, с. 357-363
12. Лукин А.С., Сторожилова М.В., Юрин Д.В. Методы анализа качества фильтрации шума на изображениях компьютерной томографии. Труды 15-й международной конференции "Цифровая обработка сигналов и её применение" (DSPA'2013), т. 2, 2013, с. 85−88.
13. Насонова А.А., Крылов А.С. Выделение сосудов на изображениях глазного дна и его оценка качества. Биотехносфера, № 3, Издательство "Политехника", 2014, с. 24−25.
3D SCIENTIFIC VISUALIZATION AND GEOMETRIC MODELING IN DIGITAL BIOMEDICINE
V.E. Turlapov, N.I. Gavrilov
Lobachevsky State University of Nizhni Novgorod, Russia
vadim.turlapov@gmail.com, gavrilov86@gmail.com
Abstract
The purpose of this paper is to review the state-of-the-art of software for three-dimensional scientific visualization, segmentation, and geometric modeling, which serves the contemporary three-dimensional digital biomedicine. The three-dimensional digital medicine is a rapidly growing industry that requires the involvement of a growing number of professionals. The importance of the topic is due to the need of developing holistic view of the area, vision of actual problems, and approaches to their solution in the scientific community.
The arsenal of libraries, software packages, and systems, which can be used in solving the actual problems, is surveyed. The focus is on the libraries of scientific visualization and 3D medical imaging, segmentation as well as geometric modeling and reconstruction. New 3D medical technologies based on these and similar libraries are presented. The possibilities of open libraries, software packages and systems for scientific visualization and segmentation, such as VTK, ITK, CMake, ParaView (all four supported by Kitware Inc.), VisIt (supported by Lawrence Livermore National Laboratory), and ITK-SNAP as well as for geometric modeling, such as CGAL, Open CASCADE, and SALOME, are characterized.
Also open software packages for segmentation and reconstruction of the data of electron microscopy (EM), such as ILASTIK, or CellBlender, and for cell modeling - Monte Carlo Cell (the last two supported by MMBioS and around community), and special package VMTK for modeling the dynamics of blood flow in the cardiovascular system are considered.
The possibilities of an open and proprietary, world and Russian modern medical 3D visualization such as Fovia HDVR®, Inobitec, Multivox3d, InVols (UNN) are presented. The problem of quality of the 3D visualization, where it is never possible to have a reference image, and the solition of this problem by synthesizing a virtual reference image by averaging a series of images with a random ray start is discussed. Pre-integration stage and research of methods in the plane of the "performance-quality" axes are found absolutely necessary for high quality code.
The possibilities, trends and depth parameterization of modern models of the human anatomy from the world's leading companies Nhumi Technologies, Visible Body, Plasticboy store, Zygote Inc. are shown. Existing products and technologies of 3D reconstruction of the patient's body via the camera and software from BodyKIT Body Labs, Itseez3d from Itseez are mentioned. Opportunities and challenges of medical technology "virtual anatomical table" and the implementation of modern Anatomage Inc., Sectra and similar functions, but as cloud services via InVols, are reviewed.
In the conclusion, we discuss the problems of digital biomedicine not yet solved by the community as well as the ways to solve them. These problems include (i) segmentation and quantification of 3D abnormalities to automate diagnosis and (ii) personalization of the parameterized model of human anatomy by extracting parameters from a personal tomography. We outline an effective approach to solving these problems by combining segmentation algorithms with geometric reconstruction in the form of parameterized models.
The novelty lies in the systemic coverage of the theme, setting actual goals and recommendations for their effective solution.
Keywords: Scientific Visualization, Geometric Modeling, 3D Digital Biomedicine, Problems, Solutions, Review
1. Gamilov T., Ivanov Yu., Kopylov P., Simakov S., Vassilevski Yu. Patient specific hemodynamic modeling after occlusion treatment in leg. Mathematical Modelling of Natural Phenomena, 2014, vol. 9, no. 6, pp. 85-97
2. Gavrilov, N. I.; Turlapov, V. E. Novel approach to development of direct volume rendering algorithms based on visualization quality assessment. Programming and Computer Software, 2014, vol. 40, no. 4, pp. 174-184
3. Engel K., Kraus M., Ertl T. High-quality pre-integrated volume rendering using hard-ware-accelerated pixel shading. Proc. of Graphics Hardware 2001, pp. 9-16
4. Knoll A., Hijazi Y., Westerteiger R., Schott M., Hansen C., Hagen H. Volume ray casting with peak finding and differential sampling. IEEE Transactions on Visualization and Computer Graphics, 2009, vol. 15, no. 6, pp. 1571–1578.
5. Lee, B., Yun, J., Seo, J., Shim, B., Shin, Y. G., & Kim, B. Fast high-quality volume ray casting with virtual samplings. IEEE Transactions on Visualization and Computer Graphics, 2010, vol. 16, no. 6, pp. 1525-1532.
6. Saito Sh., Zhou Zi-Ye, Kavan L. Computational Bodybuilding: Anatomically-based Modeling of Human Body. ACM Transactions on Graphics, 2015, vol. 34, no. 4, Article 41
7. The CGAL Project. CGAL User and Reference Manual. CGAL Editorial Board, 4.6.2 edition, 2015.
8. Yatchenko A., Krylov A. Cross-Frame Ultrasonic Color Doppler Flow Heart Image Unwrapping. Lecture Notes in Computer Science, 2015, vol. 9126, pp. 265−272
9. Yatchenko A.M., Gavrilov A.V., Boldyreva E.V., Arkhipov I.V., Grigorieva E.V., Gudkov I.M., Krylov V.V. Blood Flow Prediction and Visualization within the Aneurysm of the Middle Cerebral Artery after Surgical Treatment. Proceedings of IMTA-5, 2015, Portugal, pp. 108-113
10. Bondarev A., Galaktionov V. State-of-the-art in data visualization for CFD problems. Scientific Visualization, 2013, vol. 5, no. 4, pp. 18 – 30 [In Russian]
11. Kostin V.A., Osipov G.V. Samosoglasovannaja jelektromehanicheskaja aktivnost'' v uprugo zakreplennom volokne serdechnoj myshcy [Selfconsistent electromechanical activity elastically secured cardiac muscle fibers]. Problems of strength and plasticity, 2014, vol. 76, no 4, pp. 357-363 [In Russian]
12. Lukin A.S., Storozhilova M.V., Jurin D.V. Metody analiza kachestva fil'tracii shuma na izobrazhenijah komp'juternoj tomografii [Methods for analyzing the quality of filtering noise on CT images]. Proc. of The 15-th International Conference Digital Signal Processing and its Applications (DSPA-2013), 2013, vol. 2, с. 85−88. [In Russian]
13. Nasonova A.A., Krylov A.S. Vydelenie sosudov na izobrazhenijah glaznogo dna i ego ocenka kachestva [Isolation of blood vessels in the fundus image and quality assessment]. Biotekhnosfera, 2014, no 3, pp. 24−25. [In Russian]